这是1月31号刚出炉的一篇文章。
题目足够惊世骇俗:独特插入片段,异常相似性。
原文链接:
https://www.biorxiv.org/content/10.1101/2020.01.30.927871v1.full.pdf
题目&作者
虽然有点博眼球的嫌疑,但作者并非无名之辈,
通讯作者Bishwajit Kundu和Vivekanandan Perumal都是来自于印度理工的生物学院,是研究病毒和蛋白质的专家:
1 Abstract
摘要写得很清楚,发现四个插入与艾滋病毒高度相似的片段,不太可能像是自然界自己进化出来的。
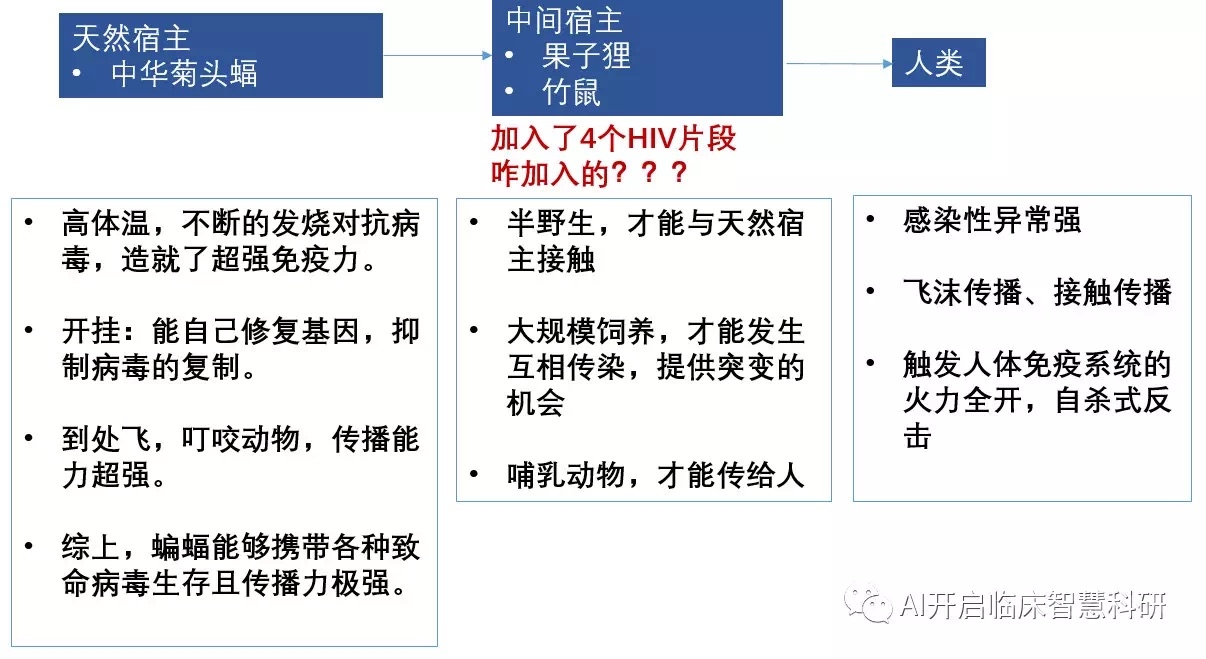
我们目前正在目睹由2019年新型冠状病毒(2019nCoV)引起的主要流行病。2019-nCoV的发展仍然难以捉摸。我们在刺突糖蛋白(S)中发现了4个插入片段,这是2019-nCoV所独有的,其他冠状病毒中没有这些插入片段。
重要的是,所有4个插入片段中的氨基酸残基均与HIV1gp120或HIV-1Gag中的氨基酸残基具有相同性或相似性。有趣的是,尽管插入片段在一级氨基酸序列上是不连续的,但2019-nCoV的3D建模表明它们会聚在一起构成受体结合位点。在2019-nCoV中发现4个独特的插入片段,这些插入片段都与HIV-1关键结构蛋白中的氨基酸残基具有同一性/相似性,这在自然界不太可能是偶然的。这项工作提供了关于2019-nCoV的未知见解,并阐明了该病毒的进化和致病性,对诊断该病毒具有重要意义。
2 Introduction
冠状病毒的刺突糖蛋白(S)被切割成两个亚基(S1和S2)。S1亚基有助于受体结合,而S2亚基促进膜融合(Bosch等,2003;Li,2016)。冠状病毒的突触糖蛋白是组织嗜性和宿主范围的重要决定因素。此外,刺突糖蛋白是疫苗开发的关键靶标(Du等人,2013)。因此,刺突蛋白代表了冠状病毒中研究最广泛的蛋白。因此,我们寻求使用计算工具研究2019-nCoV的刺突糖蛋白,以了解其进化,新特征序列和结构特征。
不了解刺突的再复习复习B站上李永乐老师的科普。
刺突就是病毒突破第一道防线细胞膜的“钥匙”,通过刺突与细胞膜的受体结合,骗过守卫才能进去细胞。
3 Methodology
我们从NCBI病毒基因组数据库(
https://www.ncbi.nlm.nih.gov/)中检索了所有可用的冠状病毒序列(n = 55),并使用了GISAID(Elbe&Buckland-Merrett,2017)[https://www.gisaid.org/]检索截至2020年1月27日的2019nCoV的所有可用全长序列(n = 28)。通过使用MUSCLE软件(Edgar,2004)和采用最近邻合并方法对所有冠状病毒基因组进行了多序列比对。在55个冠状病毒基因组中,使用MEGAX软件将所有类别的32个代表性基因组用于系统树发育(Kumar等人,2018)。发现最接近的亲属是SARS CoV。使用Multalin软件(Corpet,1988)对SARS CoV和2019-nCoV的糖蛋白区域进行比对和可视化。使用BLASTp和BLASTn将鉴定的氨基酸和核苷酸序列与整个病毒基因组数据库进行比对。通过使用MEGAX软件进行多序列比对,提出了2019-nCoV基因组的28个临床变体中核苷酸和氨基酸基序的保守性。使用SWISS-MODEL在线服务器(Biasini等,2014)生成2019-nCoV糖蛋白的三维结构,并使用PyMol对其进行标记和可视化(DeLano,2002)。
看上去比我们玩python和机器学习的专业多了。当然重点是发现与SARS非常接近。
4 Results
我们的全长冠状病毒的系统树表明,2019-nCoV与SARSCoV密切相关[图1]。
此外,其他近期研究也将2019-nCoV与SARSCoV相关联。
因此,我们将2019-nCoV的刺突糖蛋白序列与SARS CoV的刺突糖蛋白序列进行了比较(NCBI登录号:AY390556.1)。
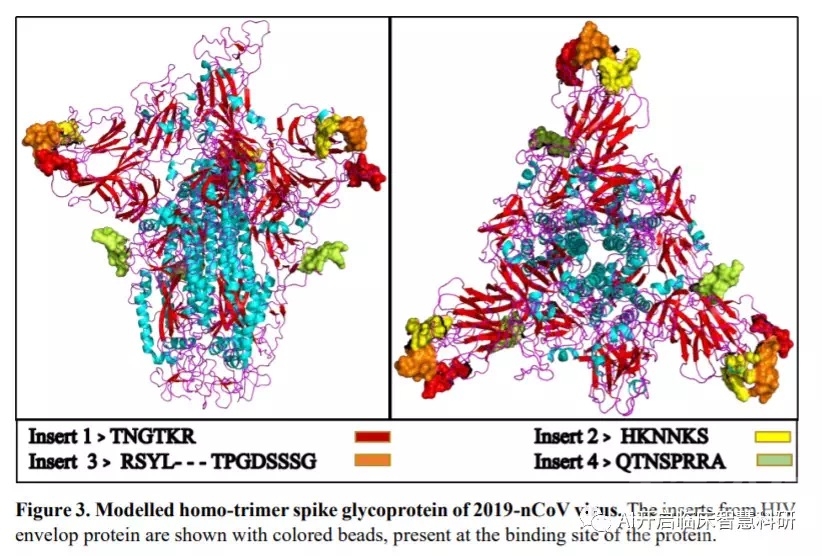
在仔细检查序列比对后,我们发现2019-nCoV峰值糖蛋白包含4个插入片段(图2)。
也就是说作者以SARS作为benchmark,观察
新冠状与SARS的区别。发现有4个插入片段不同,如下图黑色框内区域:
然后作者就将目光盯在了这四个片段上,首先发现在所有可用的2019-nCov序列分析中,这4个片段都存在(普遍性),也就是普遍存在于武汉发现的新冠状病毒中。
那就有意思了,这4个片段是哪来的呢?
为此,我们将2019-nCoV局部比对与每个插入片段作为对所有病毒基因组的查询,并考虑了100%序列覆盖率的命中。令人惊讶的是,四个插入片段中的每一个都与人类免疫缺陷病毒1(HIV-1)蛋白的短片段对齐。
表1显示了2019-nCoV中插入片段的氨基酸位置以及HIV-1 gp120和HIV-1 Gag中的相应残基。前3个插入片段(插入1、2和3)与氨基酸的短片段对齐HIV-1 gp120中的残基。插入物4与HIV-1 Gag对齐。2019-nCoV的刺突糖蛋白中的插入物1(6个氨基酸残基)和插入物2(6个氨基酸残基)与定位到HIV-1 gp120的残基100%相同。2019nCoV中的插入片段3(12个氨基酸残基)对应于带有缺口的HIV-1 gp120 [参见表1]。插入物4(8个氨基酸残基)定位于带有缺口的HIV-1 Gag。
接下来为了了解这4个插入片段的作用,科学家以SARS为研究基础,进行了建模:
基于SARS刺突糖蛋白(PDB:6ACD.1.A)的可用结构对其结构进行了建模。
建模结构的比较表明,尽管插入物1,2和3位于蛋白一级序列的非连续位置,但它们折叠后构成了识别宿主受体的糖蛋白结合位点部分(Kirchdoerfer等,2016) (图4)。
插入物1对应于2019-nCoV刺突糖蛋白中S1亚基的NTD(N末端结构域),插入物2和3对应于CTD(C末端结构域)。
插入片段4位于S1亚基的SD1(子域1)和SD2(子域2)的交界处(Ou等,2017)。
我们推测,这些插入通过在蛋白质结构中形成亲水环,可以促进或增强病毒-宿主相互作用,从而为糖蛋白结合位点提供了额外的灵活性。
根据这几天学到的粗浅的生物学知识,我理解是这4个插入片段使得病毒更加轻易地骗过细胞的守卫,更容易地进入细胞膜。这恰好解释了这次的病毒传染性为什么这么强!
而插入的4个片段都是来自于HIV病毒,这与之前报道抗艾滋病毒药物可能有效也不谋而合。
Evolutionary Analysis of 2019-nCoV
作者再次讲到了他们的研究思路,来源于动物,再传染给人,证明宿主发生了变化,从而将目光聚焦于刺突糖蛋白(S蛋白)序列。因为S蛋白是帮助病毒识别和附着宿主的表面蛋白,这些蛋白质的变化可以反映为病毒宿主特异性的变化。
然后通过比较发现与SARS高度相似,再聚焦于有差异的地方,发现了4个插入片段:“ GTNGTKR”(IS1),“ HKNNKS”(IS2),“ GDSSSG”(IS3)和“ QTNSPRRA”(IS4)
重点是这4个片段从来没有在其他冠状病毒中出现过,也不是什么新物种,而是来自于HIV-1病毒。
进一步的分析表明,HIV-1与2019-nCoV的比对序列来源于表面糖蛋白gp120(氨基酸序列位置:404-409、462-467、136150)和Gag蛋白(366-384个氨基酸)(表1)。HIV的Gag蛋白参与宿主膜的结合,病毒的包装以及病毒样颗粒的形成。Gp120通过与初级受体CD4结合在识别宿主细胞中起关键作用,这种结合诱导GP120中的结构重排,为趋化因子共受体(如CXCR4和/或CCR5)创建了高亲和力结合位点。
此外,这种结构变化可能也增加了2019-nCoV可以感染的宿主细胞范围。
也就是这插入的4个片段导致感染的可能比SARS大大大大增加了。
连作者也说:
This is startling as it is quite unlikely for a virus to have acquired such unique insertions naturally in a short duration of time.
这令人震惊,因为病毒不太可能在短时间内自然地获得这种独特的插入。
咋整,看完了这篇思路明确酣畅淋漓有理有据的论文后,我也有点不寒而栗了。
天哪,难道是在果子狸或者竹鼠等可能的中间宿主变异阶段,产生了4个HIV片段?咋加入的啊?
后记
由于不是生物专业,对内容的专业性不做评价,但就逻辑性而言,非常靠谱,一环套一环,无懈可击,very clear,值得学习!